- E’ stato stimato che di circa 5.000 – 10.000 nuove entità chimiche (NCEs), note anche come nuove entità molecolari (NMEs), scoperte in laboratorio, solo 1 riesce ad ottenere l’approvazione per il mercato [1-2-3-4-5].
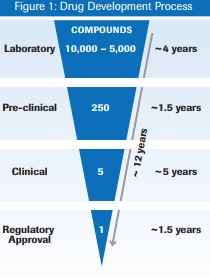
(il grafico sopra è estrapolato da un doc. pubblicato da F. Hoffmann-La Roche Ltd, 2013) [2]

- Nel 2004, la US FDA (Food and Drug Administration, organismo di farmacovigilanza degli U.S.A) stabiliva al 92% la percentuale di farmaci sperimentali che, nonostante fossero stati testati con successo nelle fasi precliniche ‘’in vivo’’ (cioè su ‘’modelli animali’’), falliva la traduzione nelle fasi cliniche (cioè su esseri umani) [6-7].
- Dati più recenti hanno rivisto questo tasso di fallimento farmacologico ad una percentuale compresa tra il 95% ed il 97% [8-9-10-11], confermato anche dagli US NIH (National Institutes of Health, l’istituzione statunitense più importante per quanto concerne la ricerca biomedica, responsabile per l’erogazione di fondi) [12]:
‘’Un nuovo farmaco, dispositivo od altro intervento, può richiedere circa 14 anni ed 1 miliardo di dollari o anche più per il suo sviluppo, con un tasso di fallimento superiore al 95%.’’ [12]
- Le ragioni principali alla base del suddetto tasso di ‘’logoramento’’ rilevato nel processo di sviluppo farmacologico (Drug Development) sono mancanza di efficacia e significativa tossicità riscontrate nei trials clinici su esseri umani [6-8-11-13-14-15], ovvero esiti del tutto divergenti con quelli rilevati nelle precedenti fasi precliniche su ‘’modelli animali’’ [6-8-11-16-17].
- Il costo connesso allo sviluppo di un nuovo agente farmacologico oscilla tra 1 e 5 miliardi di dollari statunitensi [12-18-19], altri segnalano investimenti che possono arrivare anche fino a circa 10 miliardi di dollari [20].
- Secondo uno studio eseguito dal G.A.O. (General Accounting Office degli U.S.A.), che esaminò 198 farmaci approvati tra il 1976 ed il 1985 negli Stati Uniti d’America, il 51,5% dei farmaci commercializzati manifestano ‘’Serious Adverse Effects’’ (Gravi Reazioni Avverse) nella popolazione non rilevate prima della commercializzazione [21-22-23-24].
- Uno studio più recente ha rilevato che di 222 nuovi agenti terapeutici, approvati dalla US FDA tra il 2001 ed il 2010, il 32% è stato associato ad eventi avversi [25].
- Sempre negli Stati Uniti d’America, il 2.9% dei farmaci commercializzati tra il 1975 ed il 2000 è stato ritirato e l’8.2% ha ricevuto una o più ‘’black-box warning’’ [26]. Riferiscono gli autori che hanno analizzato questi dati:
‘’Sulla base dei nostri risultati, e di quelli di altri, i medici dovrebbero evitare di utilizzare nuovi farmaci quando agenti più vecchi e di efficacia simile sono già disponibili.’’ [26]
- L’AIFA invece, ente di farmacovigilanza italiano, nel 2011 riferiva sulla situazione interna italiana quanto segue:
‘’negli ultimi 10 anni sono stati ritirati 39 farmaci’’ [27], in media 1 farmaco ritirato quasi ogni 3 mesi.
- Il Dr Allen Roses, l’allora vice-presidente di genetica presso la GlaxoSmithKline (GSK, importantissima azienda farmaceutica, tra le maggiori al mondo), ha dichiarato che almeno la metà dei pazienti a cui furono prescritti alcuni dei farmaci più costosi non godettero di alcun beneficio:
‘’la stragrande maggioranza dei farmaci, più del 90%, funziona solo nel 30 o 50% dei casi’’ [28-29-30].
- Una meta-analisi pubblicata sul JAMA nel 1998 calcolò che i morti annuali negli U.S.A. per Gravi Reazioni Avverse ai Farmaci erano 106 mila [31]. Studi più recenti hanno stimato che sono circa 128 mila l’anno le persone che muoiono sempre negli U.S.A. a causa delle Gravi Reazioni Avverse ai Farmaci (Adverse Drugs Reactions) [32], questo dato sarebbe coerente con quel trend emerso da uno studio che ha rilevato un aumento del numero delle ADRs nel corso degli anni [33-34].
- Nell’UE i morti per ADRs sono circa 197 mila all’anno [35-36-37].
- Per meglio rappresentare la magnitudo del problema ADRs, pur ammettendo che queste siano riconducibili ad errori di sorta e quindi prevenibili diciamo nel 50% od anche il 60-70% dei casi, considerando le più recenti stime [32] significherebbe che nei soli Stati Uniti d’America (cioè prendendo in esame i dati relativi ad un solo paese al mondo) si verificherebbero ogni anno oltre 600 mila casi di persone seriamente danneggiate dalle ADRs (con conseguenti ricoveri e sensibili aumenti della spesa pubblica inerenti ai costi sanitari associati), di cui oltre 38 mila sarebbero i decessi. A questo punto, si consideri altresì che nel 2013 uno studio eseguito dall’Uppsala Conflict Data Program (UCDP, Uppsala University) ha calcolato a 37.941 le vittime di guerra registrate in TUTTI i conflitti bellici in corso nel mondo nell’anno precedente [38]. In altre parole, il numero di morti da reazioni avverse ai farmaci rilevate nei soli U.S.A. ogni anno (calcolate con stime alquanto conservative) sarebbe superiore a quello delle persone decedute in TUTTE le guerre in atto nel mondo nel 2012.
- Risulta difficile calcolare una stima accurata delle Gravi Reazioni Avverse ai Farmaci, in quanto è risaputo che le stesse sono seriamente sotto-segnalate e di conseguenza sotto-riportate [26-39-40].
- Il processo di sviluppo farmacologico viene generalmente indicato come costoso ed inefficiente [19-41-42-43]:
‘’Forgiato nei primi anni del 1960, il paradigma per l’innovazione farmaceutica è rimasto praticamente invariato per quasi 50 anni. Durante un periodo in cui la maggior parte delle altre industrie basate sulla ricerca hanno adottato frequenti, e spesso radicali, modifiche ai loro processi di Ricerca e Sviluppo (R&D), il settore farmaceutico continua ad utilizzare un processo di sviluppo che è lento, inefficiente, rischioso e costoso.’’ [41]
- Una delle cause principali, se non la più rilevante, alla base di questo modello di produzione alquanto improduttivo in termini di risultati utili è da addebitare alle significative differenze inter-specie tra ‘’modelli animali’’ ed esseri umani [8-11-13-15-19-44-45-46-47-48-49-50-51-52-53].
- Gli studi preclinici condotti su animali risultano essere inadeguati, piuttosto scarsi, come ‘’sistemi di modalità predittiva’’ dell’esperienza umana (ANCHE) tossicologica di riferimento [8-11-13-15-19-44-54-55-56-57-58-59-60-61-62-63-64]:
‘’Il fatto che i candidati farmaci falliscono nei trials clinici a causa di una significativa tossicità illustra che il vecchio paradigma tossicologico sperimentale, nella fattispecie incentrato prevalentemente sull’utilizzo di animali da laboratorio per prevedere la sicurezza del farmaco per gli esseri umani, non è adeguato.’’ [59]
Questo acclarato riscontro viene confermato anche dall’O.M.S. (Organizzazione Mondiale della Sanità, WHO) che considera ‘’insufficienti’’ i test animali nel prevedere la tossicità farmacologica umana [65].
- L’attuale paradigma di ricerca biomedica e tossicologica è fortemente incentrato sul ‘’riduzionismo’’, cioè un metodo di studio che cerca di sezionare un sistema nelle sue componenti analizzandone ciascuna parte singolarmente per giungere ad una conclusione circa il sistema nel suo complesso o almeno il ruolo della singola parte [66]. Benchè il metodo riduzionista sia stato efficace nello spiegare la base chimica di numerosi processi inerenti agli organismi viventi, molti biologi ora si rendono sempre più conto che questo approccio ha raggiunto il suo limite [3-66-67-68]. Il Biologo Molecolare Marc Van Regenmortel riferisce che:
‘’I sistemi biologici sono estremamente complessi e presentano proprietà emergenti che non possono essere spiegate, o addirittura previste, studiando le loro singole parti. L’approccio riduzionista, anche se efficace nei primi giorni della biologia molecolare, sottovaluta questa complessità e pertanto ha un’influenza sempre più dannosa su molte aree della ricerca biomedica, tra cui la scoperta farmacologica e lo sviluppo di vaccini.’’ [67]
- Un approccio di ricerca sperimentale di tipo riduzionistico sarebbe l’unico possibile con il ‘’modello animale’’, in quanto attraverso la presenza di ‘’caratteri conservati tra specie’’ si potrebbe ipotizzare un’eventuale estrapolazione di dati ad un livello inter-specie. Tuttavia, animali non-umani ed esseri umani sono esempi di ‘’sistemi viventi adattativi complessi ed evoluti’’ [66] e, come tali, potrebbero reagire in maniera divergente quando ad esempio sottoposti all’azione del medesimo farmaco (come del resto dimostrato dall’elevato tasso di fallimento farmacologico rilevato nel processo di sviluppo ed imputabile, come visto, anche alle differenze tra specie). Il fatto che l’animale sia un sistema complesso, e complesso in modo diverso rispetto all’essere umano, pone seri limiti alla prospettiva riduzionista [69].
- Animali non-umani ed esseri umani possono manifestare differenti risposte agli stessi stimoli, dovute a:
- differenze genetiche;
- differenze rispetto alle mutazioni dello stesso gene (laddove una specie abbia un ortologo presente in un’altra);
- differenze rispetto alle proteine ed alla loro attività;
- differenze rispetto alla regolazione genica;
- differenze nell’espressione genica;
- differenti interazioni proteina-proteina
- differenze nelle reti genetiche;
- differenze rispetto all’organizzazione dell’organismo (esseri umani e ratti possono essere sistemi intatti, ma possono essere differentemente intatti);
- differenze nell’esposizione ambientale;
- differenze fra le storie evolutive. [66]
- Piccole differenze tra specie possono implicare enormi differenze nelle risposte davanti alla medesima perturbazione (sia essa un farmaco od una malattia) [69-70-71-72].
- Anche un singolo cambiamento amminoacidico nelle proteine tra topo ed essere umano può modificare la risposta farmacologica [72].
‘’Farmaci promettenti identificati nei modelli animali sono notoriamente difficili da tradurre per gli esseri umani a causa di piccole ma significative differenze nella fisiologia cellulare, nelle risposte genomiche ed altri fattori, come i profili lipidici.’’ [73]
‘’Piccole differenze nei modelli potrebbero portare a vaste differenze nei risultati.’’
(comportamento caotico dei sistemi viventi complessi adattativi evoluti, il cosiddetto ‘’butterfly effect’’) [74]
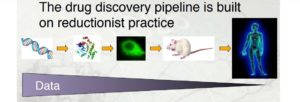

(nelle immagini sopra, tratte da: Henney, A. Opportunities and Challenges in
The Emerging Field of Synthetic Biology: Health & Medicine. A view from the pharmaceutical sector. US National Academies, OECD, Royal Society
July 9-10, 2009)
- Topi ed esseri umani hanno intrapreso percorsi evolutivi differenti separandosi da una comune specie ancestrale circa 65-85 milioni di anni fa [75-76-77].
- Gli studi genomici hanno rilevato evidenti omologie genetiche tra topi ed umani [76]. Questi studi, insieme allo sviluppo di metodi per la creazione di topi transgenici, knockout e knockin, hanno fornito impulsi considerevoli e strumenti efficaci per la ricerca con il modello murino portando ad un drastico aumento dell’uso di topi come ‘’organismi modello’’ [76]. Tuttavia, nonostante le varie analogie genetiche tra topi ed esseri umani, sono ravvisabili anche differenze significative e fondamentali. Ad esempio, le due specie variano in diversi aspetti dello sviluppo embrionale, in particolare durante la gastrulazione e l’organogenesi. Sul piano genomico, anche se la maggioranza dei geni umani e quelli del topo è ortologa, il 20% circa non ha un ortologo singolare identificabile e l’1% manca di un omologo [78].
- Topi e Ratti vengono utilizzati nella ricerca biomedica e tossicologica non tanto perché risulterebbero ‘’modelli ideali’’ per l’essere umano, ma per ragioni ‘’pratiche e di convenienza’’ [68-79-80-81].
- Anche i primati non-umani differiscono significativamente negli aspetti genetici e fisiologici dagli esseri umani [15-57-82-83-84-85-86].
- Gli scimpanzè sono ‘’i nostri parenti più stretti’’ (geneticamente parlando) e di conseguenza ci si potrebbe aspettare da loro, piuttosto che da altre specie animali di laboratorio, di avere maggiori probabilità di prevedere con una certa precisione gli esiti umani durante la ricerca preclinica. Tuttavia, nonostante la grande somiglianza tra le regioni strutturali del DNA degli scimpanzé con quello umano, differenze importanti tra le regioni regolatorie esercitano un ‘’effetto valanga’’ sul gran numero di geni strutturali [87]. Nonostante la differenza nucleotide tra scimpanzé ed esseri umani risulta essere solo dell’ 1-2%, i risultati per quanto riguarda l’espressione proteica si traducono in differenze di circa l’80% [88] con conseguenti differenze fenotipiche marcate tra le specie.
‘’Questo confronto è biologicamente significativo, perché anche una sostituzione amminoacidica potrebbe avere effetti significativi sulle differenze fenotipiche.’’ [88]
(Glazko et al. 2005)
Tali differenze si manifestano in una suscettibilità alterata ad eziologia e progressione delle malattie, differente assorbimento, distribuzione tissutale, metabolismo, escrezione degli agenti chemioterapici e differenze nella tossicità e nell’efficacia dei farmaci. Questi effetti sembrano essere responsabili dell’incapacità dimostrata nella maggior parte delle ricerche su scimpanzé di contribuire in modo sostanziale allo sviluppo di metodi efficaci nella lotta alle malattie umane [85].
- Altre specie animali di laboratorio sono ancora meno simili agli esseri umani, sia geneticamente che fenotipicamente, e quindi hanno meno probabilità di modellare accuratamente la progressione delle malattie umane o le risposte umane ad agenti farmacologici (ad esempio) [15-72-89-90-91-92].
‘’I topi sono topi e le persone sono persone. Se guardiamo al topo come modello di tutti gli aspetti della malattia per l’uomo e per le sue cure stiamo solo perdendo tempo […] Il topo ci è costato una nuova generazione di farmaci […] La maggior parte dei soldi che spendiamo negli studi clinici sulla base dei dati ottenuti sui topi è completamente sprecata […] Abbiamo avuto migliaia di studi sui topi per quanto riguarda la tubercolosi, ma nessuno di loro è mai stato utilizzato per scegliere un nuovo regime di farmaco-terapia che è riuscito nel corso degli studi clinici sull’uomo. Questo non è vero solo per la tubercolosi, ma è vero per quasi tutte le malattie. Stiamo spendendo sempre più soldi, senza ottenere potenziali farmaci utili.’’ [93]
(Dr. Clif Barry, Responsabile della Sezione Ricerca Tubercolosi, Istituto Nazionale di allergie e malattie infettive, Bethesda, Maryland, U.S.A.)
- Ad oggi, anche l’utilizzo di animali GM (geneticamente modificati) non si è rivelato risolutivo per la traduzione di validi composti farmacologici destinati al trattamento delle patologie umane. Al pari degli altri modelli animali utilizzati nelle diverse aree di ricerca, anche quelli GM vengono infatti riconosciuti come severamente limitati [72-77-94-95-96-97-98-99-100-101-102-103-104].
- In generale, i modelli animali risultano fortemente limitati e scarsamente predittivi (ANCHE) per gli studi su: diabete [90-102-105-106], cancro [15-68-77-104-107-108-109-110-111], dolore [68], immunologia [68-112-113-114-115], malattie del sistema nervoso centrale [13-68-72-77-92-116-117-118-119-120], malattie infettive come l‘HIV [68-77-82-112-121-122], studi di cancerogenesi [123-124-125-126-127-128] e teratogenesi [129-130-131-132-133].
- Tgn1412, anticorpo monoclonale anti-CD28 sviluppato per il trattamento della leucemia linfatica cronica a cellule B e dell’artrite reumatoide. Causò insufficienza multiorgano nei volontari umani a cui venne somministrato durante il trial sperimentale di fase I [134-135], ad uno dei volontari dovettero amputare le dita delle mani e dei piedi [134]. Nei test preclinici condotti su diverse specie animali, tra cui roditori e primati non-umani della specie macachi rhesus e cynomolgus [58], il Tgn1412 non manifestò segni di severa tossicità. In particolare, le scimmie utilizzate non manifestarono tossicità nella risposta al farmaco nemmeno ad un livello di dose 500 volte superiore a quella utilizzata nel trial di fase I su esseri umani [15-58]. Il macaco cynomolgus fù scelto come modello animale per la valutazione della tossicità in quanto il Tgn1412 si lega con elevata affinità ad un epitopo di sei amminoacidi su un loop extracellulare della molecola CD28 che si ritiene sia 100% omologo tra le scimmie cynomolgus e gli esseri umani [134-136]. Differenze fino al 4% circa nella sequenza amminoacidica del loop C9D del recettore CD28 sono state rilevate tra macachi rhesus ed umani [137].
‘’Come Biologo, la lezione più importante che ho imparato è che l’utilizzo dei primati non-umani per determinare la potenziale tossicità dei farmaci biologici non può essere affidabile, anche se il target molecolare è identico a quello della specie umana.’’ [138]
(Prof. Thomas Hünig, dell’Institute for Virology and Immunobiology, University of Würzburg, Germania, uno dei ricercatori che hanno preso parte allo sviluppo del Tgn1412)

‘’I modelli animali sono raramente veri modelli di malattia e possono rappresentare, nel migliore dei casi, solo alcune caratteristiche o meccanismi di malattia umana […] In alcune aree di ricerca, come per quella relativa agli agenti neuroprotettivi, il tasso di fallimento per la traduzione di farmaci efficaci dai modelli animali alla pratica clinica è stato del 100%. Per i modelli animali di dolore la situazione è analoga. Mentre sono stati proposti argomenti che suggeriscono che quanto detto sia il risultato di una scarsa metodologia e di un discutibile design del modello animale, altri concordano sul fatto che ogni specie animale rappresenta un sistema complesso evoluto e divergente che non può ritenersi predittivo per un altro (sistema complesso evoluto e divergente, ndr).’’ [139]
Jackson, C, G. (Bioscience Department, AstraZeneca R&D Charnwood, UK)
‘’Per quanto concerne l’area di studio sull’ictus, di più di 900 trattamenti neuroprotettivi che si sono dimostrati efficaci sui modelli animali utilizzati 114 sono stati testati sugli esseri umani nelle prove cliniche. Eppure, a parte due sole eccezioni, neanche uno si è dimostrato efficace per l’uso umano […] Il fatto che i modelli animali siano ancora utilizzati nella ricerca preclinica più di un decennio dopo che la loro totale mancanza di valore traslazionale è stata riportata mette in dubbio la logica del loro uso, al di là di un modo per pubblicare (i propri studi, ndr).’’ [140]
(Mullane et al. 2014)
- Il citocromo P450, responsabile del metabolismo di molti farmaci [141], sembra essersi evoluto da un unico gene ancestrale per un periodo di 1,36 miliardi di anni [142]. Ad oggi, almeno 14 famiglie di geni del CYP450 sono state identificate nei mammiferi [143]. Ogni membro di questa superfamiglia ha regioni altamente conservate nella sequenza amminoacidica. Tuttavia, nelle sequenze primarie esistono anche notevoli differenze tra specie. Anche piccole differenze nella sequenza amminoacidica possono implicare ampie differenze nella specificità del substrato [77-142-144]. Delle principali isoforme coinvolte nel metabolismo dei farmaci (CYP 1A, 2A, 2B, 2C, 2D, 2E, e 3A) quelle umane sono sostanzialmente diverse da quelle che si trovano nei roditori, nei cani e nei primati non-umani [141-145]. Tali variazioni potrebbero spiegare, almeno in parte, le notevoli divergenze nella risposta ai farmaci tra modelli animali ed esseri umani. La letteratura scientifica fornisce numerosi esempi di terapie che si sono dimostrate di successo nei modelli animali ma hanno successivamente fallito nel dimostrare di essere ugualmente performanti anche negli studi clinici:
- Circa 300 approcci farmacologici sperimentali destinati al trattamento dell’Alzheimer hanno dimostrato di essere efficaci e sicuri su modelli animali [13-146-147]. Tuttavia, ad oggi, non esiste un solo farmaco capace di fermare, rallentare, o prevenire la progressione della malattia negli esseri umani [148]. Il 99.6% dei farmaci testati con successo su animali tra il 2002 ed il 2012 ha fallito nei trials clinici sperimentali su esseri umani [149-150]. L’inadeguatezza dei modelli animali utilizzati negli studi sull’Alzheimer viene oggi riconosciuta come uno dei motivi principali alla base del mancato successo nella traduzione di valide terapie approvate per l’impiego clinico umano [13-91-116].
- La letteratura scientifica mostra che, fino ad oggi, oltre mille composti sperimentali sono stati provati con successo su modelli animali di Sclerosi Multipla [151], eppure nessuno di essi si è infine tradotto come ‘’cura decisiva’’ per la malattia umana [152]. Infatti, i modelli animali di SM vengono sempre più riconosciuti, anche in quest’area di studio, come fuorvianti per la situazione umana di riferimento [153-154-155].
- Le cose non vanno meglio per il trattamento della Sclerosi Laterale Amiotrofica [69-72-92-156]. Thomsen et al. riferiscono che, dopo mezzo secolo di prove ed oltre 150 differenti agenti terapeutici o strategie testate nei modelli preclinici animali di SLA, solo il Riluzolo è sembrato essere l’unico farmaco sviluppato capace di prolungare la sopravvivenza del paziente, anche se solo di circa 2-3 mesi, ovvero alquanto miseramente [157].
‘’Gli studi clinici basati sui modelli murini di SLA hanno ampiamente fallito, il che indica la necessità dell’esplorazione di nuovi modelli.’’ [158]
(Marchetto et al. 2011)
- Stessa frustrante situazione per il trattamento della malattia di Parkinson [159-160].
- Circa 150 farmaci sperimentali per il trattamento della sepsi sono stati testati con positività di risultato su animali, nessuno si è tradotto in una pratica clinica terapeutica efficace per l’essere umano [161].
- Circa 100 vaccini anti-HIV/AIDS hanno funzionato su modelli animali, tutti quanti hanno fallito nell’uomo in centinaia di trials clinici [82].
- Di circa mille interventi neuroprotettivi dimostrati effettivi e sicuri su animali, solo uno, il tPA (attivatore tissutale del plasminogeno, che tra l’altro per via di una serie di ragioni, tra cui il serio rischio di emorragie cerebrali, viene utilizzato solo nel 5-10% dei pazienti affetti da ictus) [162], si è tradotto dagli studi animali alla pratica clinica su esseri umani [82-163]. Un unico trattamento terapeutico per l’ictus, dopo decenni di costosissime ed estenuanti ricerche, applicabile unicamente a circa il 5-10% dei pazienti colpiti, a monte di centinaia di fallimenti.
- Circa un decennio fa, i ricercatori hanno riferito l’esistenza di 195 metodi pubblicati che hanno impedito o ritardato lo sviluppo del diabete nei modelli murini [164]. Eppure, nessuna di queste “scoperte” si è mai tradotta nella pratica clinica umana come trattamento terapeutico [106-164].
- ‘’Last but not least’’. E’ ampiamente riconosciuto tra gli addetti ai lavori della comunità scientifica, degli enti regolatori e dell’industria farmaceutica che farmaci utili, se non salva-vita, sono stati ‘’cestinati’’ prematuramente nel processo di sviluppo a causa di (fuorvianti) dati animali che non giustificavano le successive sperimentazioni umane [165-166-167].
‘’La tossicità di un farmaco è una delle ragioni più comuni per cui composti promettenti falliscono. Ma i test sugli animali, l’usuale metodo di controllo di un farmaco prima che venga provato su esseri umani, possono essere fuorvianti. Circa la metà dei farmaci che funzionano negli animali può rivelarsi tossica per le persone. Ed alcuni farmaci possono in effetti funzionare bene nelle persone, anche se non riescono negli animali, il che significa che farmaci potenzialmente importanti potrebbero essere respinti.’’ [168]
(Prof. Francis Collins, attuale Direttore degli US NIH e responsabile dell’HGP, Human Genome Project)
‘’Spesso è una questione di pura fortuna se gli esperimenti su animali portano a farmaci clinicamente utili.’’ [84-169-170]
(Dr. Bernard Brodie, Farmacologo degli US NIH)
‘’I modelli animali rappresentano niente di più che uno straordinario, e nella maggior parte dei casi irrazionale, atto di fede.’’ [98]
(Horrobin 2003)
13 novembre 2017
Alfredo Lio (socio volontario dell’associazione medico-scientifica O.S.A., in collaborazione alla L.I.M.A.V.)
Bibliografia:
[1] Li, A, P. In vitro approaches to evaluate ADMET drug properties. Curr Top Med Chem. 2004;4(7):701-6. Link: https://www.researchgate.net/publication/8668082_In_vitro_approaches_to_evaluate_ADMET_drug_properties_Curr_Top_Med_Chem_4701-706
[2] F. Hoffmann-La Roche Ltd. Understanding Clinical Trials. 2013. Link: https://moodle2.units.it/pluginfile.php/146715/mod_resource/content/1/understanding_clinical_trials.pdf
[3] Matthews, H. et al. “Omics”-Informed Drug and Biomarker Discovery: Opportunities, Challenges and Future Perspectives. Proteomes 2016, 4(3), 28; doi:10.3390/proteomes4030028. Link: http://www.mdpi.com/2227-7382/4/3/28/htm
[4] Drug Discovery and Development – Understanding the R&D Process. Innovation.org, Pharmaceutical Research and Manufacturers of America (2007). Link: http://cmidd.northwestern.edu/files/2015/10/Drug_RD_Brochure-12e7vs6.pdf
[5] Kraljevic, S. et al. EMBO Rep. 2004 Sep; 5(9): 837–842. doi: 10.1038/sj.embor.7400236. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1299137/
[6] Crawford, L. (US FDA Commissioner). Innovation or Stagnation, Challenge and Opportunity on the Critical Path to New Medical Products, 2004. Link: https://www.fda.gov/downloads/scienceresearch/specialtopics/criticalpathinitiative/criticalpathopportunitiesreports/ucm113411.pdf
[7] Huang, R. et al. Modelling the Tox21 10 K chemical profiles for in vivo toxicity prediction and mechanism characterization. Nat Commun. 2016 Jan 26;7:10425. doi: 10.1038/ncomms10425. Link: https://www.ncbi.nlm.nih.gov/pubmed/26811972
[8] Hartung, T. Look back in anger – what clinical studies tell us about preclinical work. ALTEX. 2013;30(3):275-91. Link: https://www.ncbi.nlm.nih.gov/pubmed/23861075
[9] Klinck, M, P. et al. Translational pain assessment: could natural animal models be the missing link? Pain. 2017 Sep;158(9):1633-1646. doi: 10.1097/j.pain.0000000000000978. Link: https://www.ncbi.nlm.nih.gov/pubmed/28614187
[10] Pamies, D. et al. Biological and medical applications of a brain-on-a-chip. Exp Biol Med (Maywood). 2014 Sep;239(9):1096-107. doi: 10.1177/1535370214537738. Epub 2014 Jun 9. Link: https://www.ncbi.nlm.nih.gov/pubmed/24912505
[11] Akthar, A. The flaws and human harms of animal experimentation. Camb Q Healthc Ethics. 2015 Oct;24(4):407-19. doi: 10.1017/S0963180115000079. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4594046/
[12] NCATS (US NIH). About the National Center for Advancing Translational Sciences. Link : https://ncats.nih.gov/about
[13] Langley, G, R. et al. Towards a 21st-century roadmap for biomedical research and drug discovery: consensus report and recommendations. Drug Discov Today. 2016 Oct 28. pii: S1359-6446(16)30390-7. doi: 10.1016/j.drudis.2016.10.011. Link: http://www.hsi.org/assets/pdfs/roadmap-for-research.pdf
[14] Uhl, E, W. & Warner, N, J. Mouse Models as Predictors of Human Responses: Evolutionary Medicine. Curr Pathobiol Rep. 2015;3(3):219-223. Link: https://www.ncbi.nlm.nih.gov/pubmed/26246962
[15] Coleman, R, A. Human Tissue in the Evaluation of Safety and Efficacy of New Medicines: A Viable Alternative to Animal Models? ISRN Pharm. 2011;2011:806789. doi: 10.5402/2011/806789. Epub 2011 Jul 6. Link: https://www.ncbi.nlm.nih.gov/pubmed/22389860
[16] Tagle, D, A. Tissues-on-Chips: Part II. US NIH, FY 2016 RESEARCH INITIATIVE – NCATS (2015). Link: https://ncats.nih.gov/files/council-concept-tissue-chip-6-2015.pdf
[17] Tagle, D. A. Innovative Tools for Drug Development and Disease Modeling. Novel Approaches to explore the emerging insights in the Rare Diseases Research. 2nd World Congress on Rare Diseases and Orphan Drugs, June 29-30, 2017 London, UK. Link: https://rarediseases.conferenceseries.com/europe/speaker/2017/danilo-a-tagle-national-center-for-advancing-transnational-sciencesnational-institutes-of-healthusa
[18] Khetani, S, R. et al. Microengineered liver tissues for drug testing. J Lab Autom. 2015 Jun;20(3):216-50. doi: 10.1177/2211068214566939. Epub 2015 Jan 23. Link: http://journals.sagepub.com/doi/pdf/10.1177/2211068214566939
[19] Mathur, A. et al. Human iPSC-based cardiac microphysiological system for drug screening applications. Sci Rep. 2015 Mar 9;5:8883. doi: 10.1038/srep08883. Link: https://www.ncbi.nlm.nih.gov/pubmed/25748532
[20] Kamei, K. et al. Integrated heart/cancer on a chip to reproduce the side effects of anti-cancer drugs in vitro. RSC Adv., 2017, 7, 36777-36786, DOI: 10.1039/C7RA07716E. Link: https://repository.kulib.kyoto-u.ac.jp/dspace/bitstream/2433/226624/1/c7ra07716e.pdf
[21] US G.A.O. . FDA Drug Review – Postapproval Risk, 1976-1985. April 1990. Link: http://archive.gao.gov/d24t8/141456.pdf
[22] Moore, T, J. et al. Time to act on drug safety. JAMA. 1998 May 20;279(19):1571-3. Link: https://jamanetwork.com/journals/jama/article-abstract/187529
[23] Jarernsiripornkul, N. et al. Patient reporting of potential adverse drug reactions: a methodological study. Br J Clin Pharmacol. 2002 Mar;53(3):318-25. Link: https://www.ncbi.nlm.nih.gov/pubmed/11874396
[24] Pezalla, E. Preventing adverse drug reactions in the general population. Manag Care Interface. 2005 Oct;18(10):49-52. Link: https://www.ncbi.nlm.nih.gov/pubmed/16265935
[25] Downing, N, S. et al. Postmarket Safety Events Among Novel Therapeutics Approved by the US Food and Drug Administration Between 2001 and 2010. JAMA. 2017 May 9;317(18):1854-1863. doi: 10.1001/jama.2017.5150. Link: https://www.ncbi.nlm.nih.gov/pubmed/28492899
[26] Lasser, K, E. et al. Timing of new black box warnings and withdrawals for prescription medications. JAMA. 2002 May 1;287(17):2215-20. Link: http://www.consumerwatch.org/documents/1618.pdf
[27] AIFA su studio Federanziani sulla Farmacovigilanza: non comparabili dati americani a realtà italiana. Comunicato Stampa 203, 05/07/2011. Link: http://www.agenziafarmaco.gov.it/it/content/aifa-su-studio-federanziani-sulla-farmacovigilanza-non-comparabili-dati-americani-realt%C3%A0-ita
[28] Great Britain Parliament – House of Lords: Science and Technology Committee. Genomic medicine: 2nd report of session 2008-09, Vol. 2: Evidence, Volume 2. The Stationery Office, 07 lug 2009 (pag. 511).
[29] Greek, R. et al. Animal models in an age of personalized medicine. Personalized Medicine. January 2012, Vol. 9, No. 1, Pages 47-64, DOI 10.2217/pme.11.89 (doi:10.2217/pme.11.89). Link: https://www.futuremedicine.com/doi/pdf/10.2217/pme.11.89
[30] BBC News. Drugs ‘don’t work on many people’. 8 December 2003. Link: http://news.bbc.co.uk/2/hi/health/3299945.stm
[31] Lazarou, J. et al. Incidence of adverse drug reactions in hospitalized patients: a meta-analysis of prospective studies. JAMA. 1998 Apr 15;279(15):1200-5. Link: https://www.ncbi.nlm.nih.gov/pubmed/9555760
[32] ISMP. QuarterWatch, Monitoring FDA MedWatch Reports. May 31, 2012. Link: https://www.ismp.org/quarterwatch/pdfs/2011Q4.pdf
[33] Moore, T, J. et al. Serious Adverse Drug Events Reported to the Food and Drug Administration, 1998-2005. Arch Intern Med. 2007 Sep 10;167(16):1752-9. Link: https://www.ncbi.nlm.nih.gov/pubmed/17846394
[34] Bale, A, S. et al. Correlating In Vitro Data to In Vivo Findings for Risk Assessment. ALTEX. 2014;31(1):79-90. doi: 10.14573/altex.1310011. Epub 2013 Nov 14. Link: http://www.altex.ch/resources/altex_2014_1_079_090_WR_Bale.pdf
[35] Archibald, K. et al. Barriers to the Uptake of Human-based Test Methods, and How to Overcome Them. Altern Lab Anim. 2015 Nov;43(5):301-8. Link: https://www.aerzte-gegen-tierversuche.de/images/pdf/studien/archibald_2015.pdf
[36] Anon. Strengthening pharmacovigilance to reduce adverse effects of medicines. Brussels: European Commission, 2008. Link: http://ec.europa.eu/health/files/pharmacos/pharmpack_12_2008/memo_pharmacovigiliance_december_2008_en.pdf
[37] Bouvy, J, C. et al. Epidemiology of Adverse Drug Reactions in Europe: A Review of Recent Observational Studies. Drug Saf. 2015 May;38(5):437-53. doi: 10.1007/s40264-015-0281-0. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4412588/
[38] Themnér, L. & Wallensteen, P. Armed Conflicts, 1946–2012. Journal of Peace Research, July 2013, vol. 50 no. 4 509-521. doi: 10.1177/0022343313494396. Link: http://jpr.sagepub.com/content/early/2013/06/28/0022343313494396.full.pdf
[39] Hazell, L. & Shakir, S, A. Under-reporting of adverse drug reactions : a systematic review. Drug Saf. 2006;29(5):385-96. Link: http://citeseerx.ist.psu.edu/viewdoc/download?doi=10.1.1.435.7132&rep=rep1&type=pdf
[40] Strom, B, L. Pharmacoepidemiology. John Wiley & Sons, 04 gen 2006 (pag. 153).
[41] Kaitin, K, I. Deconstructing the Drug Development Process: The New Face of Innovation. Clin Pharmacol Ther. 2010 Mar;87(3):356-61. doi: 10.1038/clpt.2009.293. Epub 2010 Feb 3. Link: https://www.ncbi.nlm.nih.gov/pubmed/20130565
[42] Sayed, N. et al. Translation of Human iPSCs: From Clinical Trial in a Dish to Precision Medicine. J Am Coll Cardiol. 2016 May 10; 67(18): 2161–2176. doi: 10.1016/j.jacc.2016.01.083. Link: http://www.onlinejacc.org/content/67/18/2161/F2 ; https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5086255/
[43] Young, S. & Sutton, K. Building an Organ on a Chip. MIT Technology Review, June 19, 2012. Link: https://www.technologyreview.com/s/427992/building-an-organ-on-a-chip/
[44] Hartung, T. Opinion versus evidence for the need to move away from animal testing. ALTEX. 2017;34(2):193-200. doi: 10.14573/altex.1703291. Link: http://www.altex.ch/resources/altex_2017_2_193_200_FFT_Hartung.pdf
[45] Esch, E, W. et al. Organs-on-chips at the frontiers of drug discovery. Nat Rev Drug Discov. 2015 Apr;14(4):248-60. doi: 10.1038/nrd4539. Epub 2015 Mar 20. Link: https://www.ncbi.nlm.nih.gov/pubmed/25792263
[46] Holmes, A, M. et al. Animal models of asthma: value, limitations and opportunities for alternative approaches. Drug Discov Today. 2011 Aug;16(15-16):659-70. doi: 10.1016/j.drudis.2011.05.014. Epub 2011 Jun 23. Link: http://csmres.co.uk/cs.public.upd/article-downloads/Holmes.pdf
[47] Dehne, E, M. et al. The ascendance of microphysiological systems to solve the drug testing dilemma. Future Sci OA. 2017 Mar 31;3(2):FSO185. doi: 10.4155/fsoa-2017-0002. eCollection 2017 Jun. Link: https://www.ncbi.nlm.nih.gov/pubmed/28670475
[48] Materne, E, M. et al. A multi-organ chip co-culture of neurospheres and liver equivalents for long-term substance testing. J Biotechnol. 2015 Jul 10;205:36-46. doi: 10.1016/j.jbiotec.2015.02.002. Epub 2015 Feb 9. Link: https://www.ncbi.nlm.nih.gov/pubmed/25678136
[49] Huh, D. et al. A human disease model of drug toxicity-induced pulmonary edema in a lung-on-a-chip microdevice. Sci Transl Med. 2012 Nov 7;4(159):159ra147. doi: 10.1126/scitranslmed.3004249. Link: https://3uyywi3w8psfe6fxn1g4tdb1-wpengine.netdna-ssl.com/wp-content/uploads/2015/06/LungChip_Sci-Transl-Med_2012.pdf
[50] Skardal, A. et al. Multi-tissue interactions in an integrated three-tissue organ-on-a-chip platform. Sci Rep. 2017 Aug 18;7(1):8837. doi: 10.1038/s41598-017-08879-x. Link: https://www.ncbi.nlm.nih.gov/pubmed/28821762
[51] Mahler, G, J. et al. Body-on-a-chip systems for animal-free toxicity testing. Altern Lab Anim. 2016 Oct;44(5):469-478. Link: https://www.ncbi.nlm.nih.gov/pubmed/27805830
[52] Langley, G. et al. Lessons from Toxicology: Developing a 21st-Century Paradigm for Medical Research. Environ Health Perspect. 2015 Nov;123(11):A268-72. doi: 10.1289/ehp.1510345. Link: https://www.ncbi.nlm.nih.gov/pubmed/26523530
[53] Low, L, A. & Tagle, D, A. Microphysiological Systems (“Organs-on-Chips”) for Drug Efficacy and Toxicity Testing. Clin Transl Sci. 2017 Jul;10(4):237-239. doi: 10.1111/cts.12444. Epub 2017 Mar 7. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5504483/
[54] Greek, R. & Menache, A. Systematic reviews of animal models: methodology versus epistemology. Int J Med Sci. 2013;10(3):206-21. doi: 10.7150/ijms.5529. Epub 2013 Jan 11. Link: https://www.ncbi.nlm.nih.gov/pubmed/23372426
[55] van Meer, P, J. et al. The ability of animal studies to detect serious post marketing adverse events is limited. Regul Toxicol Pharmacol. 2012 Dec;64(3):345-9. doi: 10.1016/j.yrtph.2012.09.002. Epub 2012 Sep 12. Link: https://www.ncbi.nlm.nih.gov/pubmed/22982732
[56] Bracken, M, B. Why animal studies are often poor predictors of human reactions to exposure. J R Soc Med. 2009 Mar;102(3):120-2. doi: 10.1258/jrsm.2008.08k033. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2746847/
[57] Passier, R. et al. Complex Tissue and Disease Modeling using hiPSCs. Cell Stem Cell. 2016 Mar 3;18(3):309-21. doi: 10.1016/j.stem.2016.02.011. Link: http://www.cell.com/cell-stem-cell/pdf/S1934-5909(16)00073-4.pdf
[58] Knight, A. Systematic Reviews of Animal Experiments Demonstrate Poor Human Clinical and Toxicological Utility. Altern Lab Anim. 2007 Dec;35(6):641-59. Link: http://www.andrewknight.info/resources/Publications/Animal-research–summaries/AK-Sys-rev-ATLA-2007-35-6.pdf
[59] Li, A, P. Human-Based in vitro Experimental Systems for the Evaluation of Human Drug Safety. In: Sahu, S, C. Hepatotoxicity: From Genomics to In Vitro and In Vivo Models. John Wiley & Sons, U.S.A., 28 feb 2008 (pag. 90).
[60] Coleman, M, D. Human Drug Metabolism: An Introduction. Second Edition. John Wiley & Sons, 30 mar 2010 (pag. 269).
[61] Jang, K, J. et al. Human kidney proximal tubule-on-a-chip for drug transport and nephrotoxicity assessment. Integr Biol (Camb). 2013 Sep;5(9):1119-29. doi: 10.1039/c3ib40049b. Link: https://www.ncbi.nlm.nih.gov/pubmed/23644926
[62] Brett, M, E. & Wood, D, K. Microsystems Models of Pathophysiolgy. In: Love, J, C. Micro- and Nanosystems for Biotechnology. John Wiley & Sons, 08 ago 2016 (pag. 217).
[63] Koutsoukas, A. et al. Predictive Toxicology: Modeling Chemical Induced Toxicological Response Combining Circular Fingerprints with Random Forest and Support Vector Machine. Front. Environ. Sci., 02 March 2016. Link: https://doi.org/10.3389/fenvs.2016.00011
[64] Hartung, T. Evolution of toxicological science: the need for change. Int. J. Risk Assessment and Management, Vol. 20, Nos. 1/2/3, 2017. Link: http://www.inderscienceonline.com/doi/pdf/10.1504/IJRAM.2017.082570
[65] Worl Health Organization. Safety of Medicines – A Guide to Detecting and Reporting Adverse Drug Reactions – Why Health Professionals Need to Take Action, 2002. Link: http://apps.who.int/medicinedocs/en/d/Jh2992e/4.html
[66] Greek, R. & Rice, M, J. Animal models and conserved processes. Theor Biol Med Model. 2012 Sep 10;9:40. doi: 10.1186/1742-4682-9-40. Link: https://www.ncbi.nlm.nih.gov/pubmed/22963674
[67] Van Regenmortel, M, H. Reductionism and complexity in molecular biology. Scientists now have the tools to unravel biological and overcome the limitations of reductionism. EMBO Rep. 2004 Nov;5(11):1016-20. Link: https://www.ncbi.nlm.nih.gov/pubmed/15520799
[68] Green, S, B. Can animal data translate to innovations necessary for a new era of patient-centred and individualised healthcare? Bias in preclinical animal research. BMC Med Ethics. 2015 Jul 28;16:53. doi: 10.1186/s12910-015-0043-7. Link: https://www.ncbi.nlm.nih.gov/pubmed/26215508
[69] Greek, R. & Hansen, L, A. Questions regarding the predictive value of one evolved complex adaptive system for a second: exemplified by the SOD1 mouse. Prog Biophys Mol Biol. 2013 Nov;113(2):231-53. doi: 10.1016/j.pbiomolbio.2013.06.002. Epub 2013 Jun 20. Link: https://www.ncbi.nlm.nih.gov/pubmed/23791779
[70] Barry, S, M. et al. (2014). Trial, Error, and Breakthrough: A Review of HIV Vaccine Development. J AIDS Clin Res 5: 359. doi:10.4172/2155-6113.1000359. Link: https://www.omicsonline.org/open-access/trial-error-and-breakthrough-a-review-of-hiv-vaccine-development-2155-6113.1000359.php?aid=32206
[71] Sausville, E, A. Preclinical Models for Anticancer Drug Development. In: Garrett-Mayer, E. Principles of Anticancer Drug Development. Springer Science & Business Media, 29 dic 2010 (pag. 107).
[72] Haston, K, M. & Finkbeiner, S. Clinical Trials in a Dish: The Potential of Pluripotent Stem Cells to Develop Therapies for Neurodegenerative Diseases. Annu Rev Pharmacol Toxicol. 2016;56:489-510. doi: 10.1146/annurev-pharmtox-010715-103548. Epub 2015 Oct 28. Link: https://www.ncbi.nlm.nih.gov/pubmed/26514199
[73] Moraes, C. et al. On being the right size: scaling effects in designing a human-on-a-chip. Integr Biol (Camb). 2013 Sep;5(9):1149-61. doi: 10.1039/c3ib40040a. Link: https://www.ncbi.nlm.nih.gov/pubmed/23925524
[74] Mergenthaler, P. & Meisel, A. Animal models: value and translational problems. In: Wehling, M. Principles of Translational Science in Medicine: From Bench to Bedside. Academic Press, 02 apr 2015 (pag. 86)
[75] Mestas, J. & Hughes, C, C. Of mice and not men: differences between mouse and human immunology. J Immunol. 2004 Mar 1;172(5):2731-8. Link: https://www.ncbi.nlm.nih.gov/pubmed/14978070
[76] Perlman, R, L. Mouse models of human disease: An evolutionary perspective. Evol Med Public Health. 2016 May 21;2016(1):170-6. doi: 10.1093/emph/eow014. Print 2016. Link: https://www.ncbi.nlm.nih.gov/pubmed/27121451
[77] Penco, S, Venco, E, Lio, A. Lost in translation: the need for better tools. Altern Lab Anim. 2014 Sep;42(4):P41-5. Link: http://pilas.org.uk/lost-in-translation-the-need-for-better-tools/
[78] Avior, Y. et al. Pluripotent stem cells in disease modelling and drug discovery. Nat Rev Mol Cell Biol. 2016 Mar;17(3):170-82. doi: 10.1038/nrm.2015.27. Epub 2016 Jan 28. Link: http://dls.ym.edu.tw/course/hb/doc/lecture20-iPS%20and%20drug%20discovery%202016.pdf
[79] Hartung, T. Lessons Learned from Alternative Methods and their Validation for a New Toxicology in the 21st Century. J Toxicol Environ Health B Crit Rev. 2010 Feb;13(2-4):277-90. doi: 10.1080/10937404.2010.483945. Link: https://www.researchgate.net/publication/44699093_Lessons_Learned_from_Alternative_Methods_and_their_Validation_for_a_New_Toxicology_in_the_21st_Century
[80] Hartung, T. From alternative methods to a new toxicology. Eur J Pharm Biopharm. 2011 Apr;77(3):338-49. doi: 10.1016/j.ejpb.2010.12.027. Epub 2010 Dec 30. Link: https://www.ncbi.nlm.nih.gov/pubmed/21195172
[81] Radermacher, P. & Haouzi, P. A mouse is not a rat is not a man: species-specific metabolic responses to sepsis – a nail in the coffin of murine models for critical care research? Intensive Care Med Exp. 2013 Dec;1(1):26. doi: 10.1186/2197-425X-1-7. Epub 2013 Oct 29. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4796700/
[82] Bailey, J. Monkey-based research on human disease: the implications of genetic differences. Altern Lab Anim. 2014 Nov;42(5):287-317. Link: https://www.ncbi.nlm.nih.gov/pubmed/25413291
[83] van Meer, P, J. et al. The value of non-human primates in the development of monoclonal antibodies. Nat Biotechnol. 2013 Oct;31(10):882-3. doi: 10.1038/nbt.2709. Link: http://m.tipharma.com/fileadmin/user_upload/Theses/PDF/Peter_van_Meer_T6-301.pdf#page=40
[84] Hartung, T. Food for thought… on animal tests. ALTEX. 2008;25(1):3-16. Link: http://www.altex.ch/resources/altex_2008_1_3_9_FFT_HartungE.pdf
[85] Knight, A. The poor contribution of chimpanzee experiments to biomedical progress. J Appl Anim Welf Sci. 2007;10(4):281-308. Link: http://www.andrewknight.info/resources/Publications/Animal-research–medical-applications/AK-Chimp-expts-JAAWS-2007-10-4.pdf
[86] Pound, P. & Bracken, M, B. Is animal research sufficiently evidence based to be a cornerstone of biomedical research? BMJ. 2014 May 30;348:g3387. doi: 10.1136/bmj.g3387. Link: https://aerzte-gegen-tierversuche.de/images/pdf/studien/bmj_pound_2014.pdf
[87] Bailey, J. Non-human primates in medical research and drug development: a critical review. Biog Amines 2005; 19(4-6): 235–55. Link: http://animalstudiesrepository.org/cgi/viewcontent.cgi?article=1029&context=acwp_lab
[88] Glazko, G. et al. Eighty percent of proteins are different between humans and chimpanzees. Gene. 2005 Feb 14;346:215-9. Link: http://test.scripts.psu.edu/users/n/x/nxm2/2005%20Publications/2005-glazko-etal.pdf
[89] Coggins, C, R. A minireview of chronic animal inhalation studies with mainstream cigarette smoke. Inhal Toxicol. 2002 Oct;14(10):991-1002. Link: https://www.ncbi.nlm.nih.gov/pubmed/12396407
[90] Wang, B. et al. Leptin- and leptin receptor-deficient rodent models: relevance for human type 2 diabetes. Curr Diabetes Rev. 2014 Mar;10(2):131-45. Link: https://www.ncbi.nlm.nih.gov/pubmed/24809394
[91] Pistollato, F. et al. Alzheimer disease research in the 21st century: past and current failures, new perspectives and funding priorities. Oncotarget. 2016 Jun 28;7(26):38999-39016. doi: 10.18632/oncotarget.9175. Link: https://www.ncbi.nlm.nih.gov/pubmed/27229915
[92] Clerc, P. et al. A look into the future of ALS research. Drug Discov Today. 2016 Jun;21(6):939-49. doi: 10.1016/j.drudis.2016.02.002. Epub 2016 Feb 6. Link: http://csmres.co.uk/cs.public.upd/article-downloads/A-look-into-the-future-of-ALS-research.pdf
[93] Engber, D. The Mouse Trap – The dangers of using one lab animal to study every disease. Slate, Nov. 19, 2011. Link: http://www.slate.com/articles/health_and_science/the_mouse_trap/2011/11/lab_mice_are_they_limiting_our_understanding_of_human_disease_.html
[94] Bhogal, N. Genetically altered mice, man and medicine. J Appl Biomed, 2008; 6(2) 47–56. Link: http://jab.zsf.jcu.cz//6_2/bhogal.pdf
[95] Lin, J, H. Applications and limitations of genetically modified mouse models in drug discovery and development. Curr Drug Metab. 2008 Jun;9(5):419-38. Link: https://www.ncbi.nlm.nih.gov/pubmed/18537578
[96] Geerts, H. Of mice and men: bridging the translational disconnect in CNS drug discovery. CNS Drugs. 2009 Nov;23(11):915-26. doi: 10.2165/11310890-000000000-00000. Link: http://www.in-silico-biosciences.com/intro/ISB_Geerts2009_OfMiceAndMen.pdf
[97] Stingl, L. et al. 20 years of hypertension research using genetically modified animals: no clinically promising approaches in sight. ALTEX. 2009;26(1):41-51. Link: https://www.ncbi.nlm.nih.gov/pubmed/19326032
[98] Horrobin, D, F. Modern biomedical research: an internally self-consistent universe with little contact with medical reality? Nat Rev Drug Discov. 2003 Feb;2(2):151-4. Link: http://www.nature.com/nrd/journal/v2/n2/full/nrd1012.html?foxtrotcallback=true
[99] Bhogal, N. & Combes, R. The relevance of genetically altered mouse models of human disease. Altern Lab Anim. 2006 Aug;34(4):429-54. Link: https://pdfs.semanticscholar.org/af3b/ed7bbfb99243c603e0bdfed85427826063bd.pdf
[100] Giri, S. & Bader, A. A low-cost, high-quality new drug discovery process using patient-derived induced pluripotent stem cells. Drug Discov Today. 2015 Jan;20(1):37-49. doi: 10.1016/j.drudis.2014.10.011. Epub 2014 Nov 15. Link: https://www.ncbi.nlm.nih.gov/pubmed/25448756
[101] Langley, G, R. Considering a new paradigm for Alzheimer’s disease research. Drug Discov Today. 2014 Mar 21. pii: S1359-6446(14)00090-7. doi: 10.1016/j.drudis.2014.03.013. Link: http://animalstudiesrepository.org/cgi/viewcontent.cgi?article=1052&context=acwp_arte
[102] Bunner, A, E. et al. Knockout mouse models of insulin signaling: Relevance past and future. World J Diabetes. 2014 Apr 15;5(2):146-59. doi: 10.4239/wjd.v5.i2.146. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3990311/
[103] Benam, K, H. et al. Engineered in vitro disease models. Annu Rev Pathol. 2015;10:195-262. doi: 10.1146/annurev-pathol-012414-040418. Link: https://pdfs.semanticscholar.org/9cca/cf7a73f76aea974853a2f36a16cb183bb068.pdf
[104] Enna, S, J. & Williams, M. Defining the role of pharmacology in the emerging world of translational research. Adv Pharmacol. 2009;57:1-30. Link: https://www.ncbi.nlm.nih.gov/pubmed/20230758
[105] Lai, M. et al. You are what you eat, or are you? The challenges of translating high-fat-fed rodents to human obesity and diabetes. Nutr Diabetes. 2014 Sep 8;4:e135. doi: 10.1038/nutd.2014.30. Link: http://www.nature.com/nutd/journal/v4/n9/full/nutd201430a.html
[106] Chandrasekera, P, C. & Pippin, J, J. Of rodents and men: species-specific glucose regulation and type 2 diabetes research. ALTEX. 2014;31(2):157-76. Link: http://www.altex.ch/resources/altex_2014_2_157_176_Chandrasekera.pdf
[107] Mak, I, W. et al. Lost in translation: animal models and clinical trials in cancer treatment. Am J Transl Res. 2014 Jan 15;6(2):114-8. eCollection 2014. Link: https://www.ncbi.nlm.nih.gov/pubmed/24489990
[108] Charbe, N. et al. Three-dimensional bio-printing: A new frontier in oncology research. World J Clin Oncol. 2017 Feb 10;8(1):21-36. doi: 10.5306/wjco.v8.i1.21. Link: https://www.ncbi.nlm.nih.gov/pubmed/28246583
[109] Jackson, S, J. & Thomas, G, J. Human tissue models in cancer research: looking beyond the mouse. Dis Model Mech. 2017 Aug 1;10(8):939-942. doi: 10.1242/dmm.031260. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5560067/
[110] Constant, S. et al. Advanced Human In vitro Models for the Discovery and Development of Lung Cancer Therapies. In: Vallisuta, O. & Olimat, S. Drug Discovery and Development – From Molecules to Medicine. InTech, June 03, 2015. DOI: 10.5772/60606. Link: https://cdn.intechopen.com/pdfs-wm/48483.pdf
[111] NC3Rs. Workshop report: Minimising animal use in preclinical oncology research (2016). Link: https://www.nc3rs.org.uk/news/workshop-report-published-minimising-animal-use-preclinical-oncology-research
[112] Koff, W, C. et al. Accelerating next-generation vaccine development for global disease prevention. Science. 2013 May 31;340(6136):1232910. doi: 10.1126/science.1232910. Link: https://www.ncbi.nlm.nih.gov/pubmed/23723240
[113] Brodin, P. & Davis, M, M. Human immune system variation. Nat Rev Immunol. 2017 Jan;17(1):21-29. doi: 10.1038/nri.2016.125. Epub 2016 Dec 5. Link: https://www.ncbi.nlm.nih.gov/pubmed/27916977
[114] Davis, M, M. A prescription for human immunology. Immunity. 2008 Dec 19;29(6):835-8. doi: 10.1016/j.immuni.2008.12.003. Link: https://www.ncbi.nlm.nih.gov/pubmed/19100694
[115] Khanna, R. & Burrows, S, R. Human immunology: a case for the ascent of non-furry immunology. Immunol Cell Biol. 2011 Mar;89(3):330-1. doi: 10.1038/icb.2010.173. https://www.nature.com/icb/journal/v89/n3/full/icb2010173a.html
[116] Choi, S, H. et al. 3D culture models of Alzheimer’s disease: a road map to a “cure-in-a-dish”. Mol Neurodegener. 2016 Dec 9;11(1):75. doi: 10.1186/s13024-016-0139-7. Link: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5148918/
[117] Papassotiropoulos, A. & de Quervain, D, J. Failed drug discovery in psychiatry: time for human genome-guided solutions. Trends Cogn Sci. 2015 Apr;19(4):183-7. doi: 10.1016/j.tics.2015.02.002. Epub 2015 Feb 27. Link: http://www.geneguide.com/GeneGuide_files/PDF.pdf
[118] Pistollato, F. et al. A human-based integrated framework for Alzheimer’s disease research. J Alzheimers Dis. 2015;47(4):857-68. doi: 10.3233/JAD-150281. Link: https://www.ncbi.nlm.nih.gov/pubmed/26401764
[119] Hyman, S, E. Revolution stalled. Sci Transl Med. 2012 Oct 10;4(155):155cm11. doi: 10.1126/scitranslmed.3003142. Link: http://stm.sciencemag.org/content/scitransmed/4/155/155cm11.full.pdf
[120] Wang, S, J. et al. Adaptive design clinical trials and trial logistics models in CNS drug development. Eur Neuropsychopharmacol. 2011 Feb;21(2):159-66. doi: 10.1016/j.euroneuro.2010.09.003. Link: http://www.ncbi.nlm.nih.gov/pubmed/20933373
[121] Greek, R. Animal Models and the Development of an HIV Vaccine. J AIDS Clinic Res 2012, S8. doi: 10.4172/2155-6113.S8-001. Link: http://omicsonline.org/animal-models-and-the-development-of-an-hiv-vaccine-2155-6113.S8-001.pdf
[122] Barh, D. et al. OMICS: Biomedical Perspectives and Applications. CRC Press, 21 nov 2011. (pag. 504)
[123] Knight, A. et al. Animal carcinogenicity studies: 1. Poor human predictivity. Altern Lab Anim. 2006 Feb;34(1):19-27. Link: http://animalstudiesrepository.org/cgi/viewcontent.cgi?article=1000&context=acwp_arte
[124] Knight, A. et al. Animal carcinogenicity studies: 2. Obstacles to extrapolation of data to humans. Altern Lab Anim. 2006 Feb;34(1):29-38. Link: http://animalstudiesrepository.org/cgi/viewcontent.cgi?article=1090&context=acwp_arte
[125] Caiment, F. et al. Assessing compound carcinogenicity in vitro using connectivity mapping. Carcinogenesis. 2014 Jan;35(1):201-7. doi: 10.1093/carcin/bgt278. Epub 2013 Aug 12. Link: https://www.ncbi.nlm.nih.gov/pubmed/23940306
[126] Basketter, D, A. et al. A Roadmap for the Development of Alternative (Non-Animal) Methods for Carcinogenicity Testing. In: A roadmap for the development of alternative (non-animal) methods for systemic toxicity testing – t4 report*. ALTEX. 2012;29(1):3-91. Link: https://www.ncbi.nlm.nih.gov/pubmed/22307314
[127] Ennever, F, K. & Lave, L, B. Implications of the lack of accuracy of the lifetime rodent bioassay for predicting human carcinogenicity. Regul Toxicol Pharmacol. 2003 Aug;38(1):52-7. Link: https://pdfs.semanticscholar.org/90b9/f63a8ddfd423265880dac7f3398bd386b706.pdf
[128] Ward, J, M. The Two-Year Rodent Carcinogenesis Bioassay — Will It Survive? J Toxicol Pathol 2007; 20: 13-19. Link: http://globalvetpathology.com/images/jmward_Two-Year_Rodent_Bioassay.pdf
[129] Cartwright, A, C. & Matthews, B, R. International Pharmaceutical Product Registration, Second Edition (from international experts in the pharmaceutical industry, academia, and regulatory bodies). CRC Press, 19 apr 2016 (pag. 624).
[130] Briggs, G, G. et al. Should pregnant women be included in phase IV clinical drug trials? Am J Obstet Gynecol. 2015 Dec;213(6):810-5. doi: 10.1016/j.ajog.2015.05.047. Epub 2015 May 23. Link: https://www.ncbi.nlm.nih.gov/pubmed/26008178
[131] Bailey, J. et al. The future of teratology research is in vitro. Biogenic Amines, Vol. 19, No. 2, pp. 97–145 (2005). Link: http://animalstudiesrepository.org/cgi/viewcontent.cgi?article=1023&context=acwp_arte
[132] Bremer, S. et al. The development of new concepts for assessing reproductive toxicity applicable to large scale toxicological programmes. Curr Pharm Des. 2007;13(29):3047-58. Link: http://animalstudiesrepository.org/cgi/viewcontent.cgi?article=1047&context=acwp_arte
[133] Salem, H, Dorsey, R, Carmany, D, Hartung, T. In Vivo, In Vitro, and Stem Cells Technologies to Predict Human Pharmacology and Toxicology. In: Murphy, S, V. & Atala, A. Regenerative Medicine Technology: On-a-Chip Applications for Disease Modeling, Drug Discovery and Personalized Medicine. CRC Press, 30 nov 2016 (Cap. 17.1)
[134] Danilenko, D, M. & Wang, H. The yin and yang of immunomodulatory biologics: assessing the delicate balance between benefit and risk. Toxicol Pathol. 2012;40(2):272-87. doi: 10.1177/0192623311430237. Epub 2012 Jan 5. Link: http://journals.sagepub.com/doi/pdf/10.1177/0192623311430237
[135] L’ orrore del Tgn1412. La Repubblica.it, 7 maggio 2006. Link: http://ricerca.repubblica.it/repubblica/archivio/repubblica/2006/05/07/orrore-del-tgn1412.html
[136] Hünig, T. The storm has cleared: lessons from the CD28 superagonist TGN1412 trial. Nat Rev Immunol. 2012 Apr 10;12(5):317-8. doi: 10.1038/nri3192. Link: https://www.researchgate.net/profile/Thomas_Huenig/publication/223969275_The_storm_has_cleared_Lessons_from_the_CD28_superagonist_TGN1412_trial/links/5770dc6508ae10de639c1564/The-storm-has-cleared-Lessons-from-the-CD28-superagonist-TGN1412-trial.pdf
[137] Hansen, S. & Leslie, R, G. TGN1412: Scrutinizing preclinical trials of antibody-based medicines. Nature. 2006 May 18;441(7091):282. Link: https://www.nature.com/nature/journal/v441/n7091/full/441282a.html
[138] Hünig, T. The rise and fall of the CD28 superagonist TGN1412 and its return as TAB08: a personal account. FEBS J. 2016 Sep;283(18):3325-34. doi: 10.1111/febs.13754. Epub 2016 Jun 6. Link: http://onlinelibrary.wiley.com/doi/10.1111/febs.13754/epdf
[139] Jackson, C, G. Target Selection and Validation in Drug Discovery. Top Med Chem (2014) 11: 1-72. DOI: 10.1007/7335_2014_59. In: Empfield, J, R. & Clark, M, P. Reducing Drug Attrition. Topics in Medicinal Chemistry, vol. 11 (pag. 13). Springer, 27 nov. 2014.
[140] Mullane, K. et al. Translational paradigms in pharmacology and drug discovery. Biochem Pharmacol. 2014 Jan 1;87(1):189-210. doi: 10.1016/j.bcp.2013.10.019. Epub 2013 Oct 30. Link: https://www.ncbi.nlm.nih.gov/pubmed/24184503
[141] Bailey, J. et al. Predicting human drug toxicity and safety via animal tests: can any one species predict drug toxicity in any other, and do monkeys help? Altern Lab Anim. 2015 Dec;43(6):393-403. Link: http://animalstudiesrepository.org/cgi/viewcontent.cgi?article=1023&context=acwp_lab
[142] Lin, J, H. & Lu, A, Y. Role of Pharmacokinetics and Metabolism in Drug Discovery and Development. Pharmacol Rev. 1997 Dec;49(4):403-49. Link: http://pharmrev.aspetjournals.org/content/pharmrev/49/4/403.full.pdf
[143] Nelson, D, R. et al. P450 superfamily: update on new sequences, gene mapping, accession numbers and nomenclature. Pharmacogenetics. 1996 Feb;6(1):1-42. Link: https://www.ncbi.nlm.nih.gov/pubmed/8845856
[144] Lindbergh, R, L. & Negishi, M. Alteration of mouse cytochrome P450coh substrate specificity by mutation of a single amino-acid residue. Nature. 1989 Jun 22;339(6226):632-4. Link: https://www.ncbi.nlm.nih.gov/pubmed/2733794
[145] Li, A, P. Evaluation of Adverse Drug Properties with Cryopreserved Human Hepatocytes and the Integrated Discrete Multiple Organ Co-culture (IdMOCTM) System. Toxicol Res. 2015 Jun; 31(2): 137–149. doi: 10.5487/TR.2015.31.2.137. Link: http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4505344/
[146] Coppola, G. The OMICs: Applications in Neuroscience. OUP USA, 2014 (pag. 300).
[147] Geerts, H. et al. Big data to smart data in Alzheimer’s disease: The brain health modeling initiative to foster actionable knowledge. Alzheimers Dement. 2016 Sep;12(9):1014-21. doi: 10.1016/j.jalz.2016.04.008. Epub 2016 May 26. Link: http://www.sciencedirect.com/science/article/pii/S1552526016302461
[148] Mertens, J. et al. APP Processing in Human Pluripotent Stem Cell-Derived Neurons Is Resistant to NSAID-Based γ-Secretase Modulation. Stem Cell Reports. 2013 Dec 5;1(6):491-8. doi: 10.1016/j.stemcr.2013.10.011. eCollection 2013. Link: https://www.ncbi.nlm.nih.gov/pubmed/24371804
[149] Cummings, J, L. et al. Alzheimer’s disease drug-development pipeline: few candidates, frequent failures. Alzheimers Res Ther. 2014 Jul 3;6(4):37. doi: 10.1186/alzrt269. eCollection 2014. Link: https://www.ncbi.nlm.nih.gov/pubmed/25024750
[150] Lin, A, L. Alzheimer’s disease: Treatment failures and future directions. Adjacent Open Access, 05/12/16. Link: https://www.openaccessgovernment.org/alzheimers-disease-treatment-failures-future-directions/30355/
[151] Baker, D. & Amor, S. Mouse models of multiple sclerosis: lost in translation? Curr Pharm Des. 2015;21(18):2440-52. Link: https://www.ncbi.nlm.nih.gov/pubmed/25777759
[152] Pippin, J, J. Animal research in medical science: seeking a convergence of science, medicine, and animal law. South Texas Law Review 2013; 54: 469-511. Link: http://faculty.smu.edu/jkazez/ar13/pippin.pdf
[153] Sriram, S. & Steiner, I. Experimental allergic encephalomyelitis: a misleading model of multiple sclerosis. Ann Neurol. 2005 Dec;58(6):939-45. Link: http://www.direct-ms.org/sites/default/files/EAEcritque.pdf
[154] Behan, P, O. & Chaudhuri, A. The sad plight of multiple sclerosis research (low on fact, high on fiction): critical data to support it being a neurocristopathy. Inflammopharmacology. 2010 Dec;18(6):265-90. Link: http://www.ncbi.nlm.nih.gov/pubmed/20862553
[155] Rice, J. Animal models: Not close enough. Nature. 2012 Apr 12;484(7393):S9. Link: http://www.nature.com/nature/journal/v484/n7393_supp/full/nature11102.html
[156] Schnabel, J. Neuroscience: Standard Model. Nature. 2008 Aug 7;454(7205):682-5. doi: 10.1038/454682a. Link: https://www.nature.com/news/2008/080806/full/454682a.html
[157] Thomsen, G, M. et al. The past, present and future of stem cell clinical trials for ALS. Exp Neurol. 2014 Dec;262 Pt B:127-37. doi: 10.1016/j.expneurol.2014.02.021. Epub 2014 Mar 6. Link: http://www.sciencedirect.com/science/article/pii/S0014488614000739/pdfft?md5=7a57ecda8be71758d064ebd01aae24f9&pid=1-s2.0-S0014488614000739-main.pdf
[158] Marchetto, M, C. et al. Induced pluripotent stem cells (iPSCs) and neurological disease modeling: progress and promises. Hum Mol Genet. 2011 Oct 15;20(R2):R109-15. doi: 10.1093/hmg/ddr336. Epub 2011 Aug 9. Link: https://www.ncbi.nlm.nih.gov/pubmed/21828073
[159] Athauda, D. & Foltynie, T. The ongoing pursuit of neuroprotective therapies in Parkinson disease. Nat Rev Neurol. 2015 Jan;11(1):25-40. doi: 10.1038/nrneurol.2014.226. Epub 2014 Dec 2. Link: https://www.ncbi.nlm.nih.gov/pubmed/25447485
[160] Potashkin, J, A. et al. Limitations of Animal Models of Parkinson’s Disease. Parkinsons Dis. 2010 Dec 20;2011:658083. doi: 10.4061/2011/658083. Link: https://www.ncbi.nlm.nih.gov/pubmed/21209719
[161] Seok, J. et al. Genomic responses in mouse models poorly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 2013 Feb 26;110(9):3507-12. doi: 10.1073/pnas.1222878110. Epub 2013 Feb 11. Link: https://www.ncbi.nlm.nih.gov/pubmed/23401516
[162] Corbett, D. et al. Lost in translation: rethinking approaches to stroke recovery. Prog Brain Res. 2015;218:413-34. doi: 10.1016/bs.pbr.2014.12.002. Link: https://www.researchgate.net/publication/276267106_Lost_in_translation_Rethinking_approaches_to_stroke_recovery
[163] Akhlaq A. Farooqui. Neurochemical Aspects of Neurotraumatic and Neurodegenerative Diseases. Springer Science & Business Media, 2010 (pag. 72).
[164] Roep, B, O. et al. Satisfaction (not) guaranteed: re-evaluating the use of animal models of type 1 diabetes. Nat Rev Immunol. 2004 Dec;4(12):989-97. doi: 10.1038/nri1502. Link: https://www.ncbi.nlm.nih.gov/pubmed/15573133
[165] Chapin, R, E. et al. Assuring safety without animal testing: the case for the human testis in vitro. Reprod Toxicol. 2013 Aug;39:63-8. doi: 10.1016/j.reprotox.2013.04.004. Epub 2013 Apr 21. Link: https://s3.amazonaws.com/academia.edu.documents/46551627/Assuring_Safety_Without_Animal_Testing_T20160616-13474-m1mx2b.pdf?AWSAccessKeyId=AKIAIWOWYYGZ2Y53UL3A&Expires=1507928222&Signature=4llRLZlAc6mvHpfugY3tBvHpneI%3D&response-content-disposition=inline%3B%20filename%3DAssuring_safety_without_animal_testing_T.pdf
[166] Pankevich, D, E. et al. Improving and Accelerating Drug Development for Nervous System Disorders. Neuron. 2014 Nov 5;84(3):546-53. doi: 10.1016/j.neuron.2014.10.007. Epub 2014 Nov 5. Link: https://www.ncbi.nlm.nih.gov/pubmed/25442933
[167] Institute of Medicine. 2009. Assessing and Predicting Kidney Safety, in: Accelerating the Development of Biomarkers for Drug Safety: Workshop Summary. Washington, DC: The National Academies Press. Link: https://www.ncbi.nlm.nih.gov/books/NBK32719/
[168] U.S. to develop chip that tests if a drug is toxic. Reuters, October 6, 2011. Link: http://www.reuters.com/article/us-drugs-chip/u-s-to-develop-chip-that-tests-if-a-drug-is-toxic-idUSTRE78F5KX20110916
[169] Brodie, B. Of mice, microsomes and man. Pharmacologist 6, 12-26 (1964).
[170] Lin, J, H. Species similarities and differences in pharmacokinetics. Drug Metab Dispos. 1995 Oct;23(10):1008-21. Link: http://citeseerx.ist.psu.edu/viewdoc/download?doi=10.1.1.842.9148&rep=rep1&type=pdf
0 comments